library(RPostgres)
library(dplyr)
library(aws.s3)
library(ggplot2)
library(raster)
library(sf)
library(janitor)
library(knitr)
# Pour avoir les noms de dates en français
invisible(Sys.setlocale("LC_ALL", "fr_FR.UTF-8"))
options(knitr.kable.NA = "")
cnx <- dbConnect(Postgres(),
user = Sys.getenv("USER_POSTGRESQL"),
password = Sys.getenv("PASS_POSTGRESQL"),
host = Sys.getenv("HOST_POSTGRESQL"),
dbname = "defaultdb",
port = 5432,
check_interrupts = TRUE)3 Cultures et prévisions climatiques
RPG, prévisions, cultures
Le changement climatique aura des changements importants sur les cultures en France, en particulier à cause de la diminution des précipitations dans certaines régions au cours des années à venir. Dans cette application, on souhaite identifier les cultures avec le niveau de risque le plus élevé.
3.1 Données
Le RPG recense les parcelles déclarées à la PAC par les agriculteurs, leurs informations graphiques et leur culture principale. Ces données sont mises à disposition dans une base de données PostgreSQL.
Le projet Drias a pour vocation de mettre à disposition des projections climatiques réalisées dans les laboratoires français de modélisation du climat (IPSL, CERFACS, CNRM). En particulier, nous disposons de projections locales du modèle CNRM-CM5 / ALADIN63 / correction ADAMONT. Ces données sont aussi mises à disposition sur PostgreSQL.
3.2 Visualisations
On initialise une nouvelle fois la connexion à PostgreSQL comme expliqué à la fin de l’application 0.
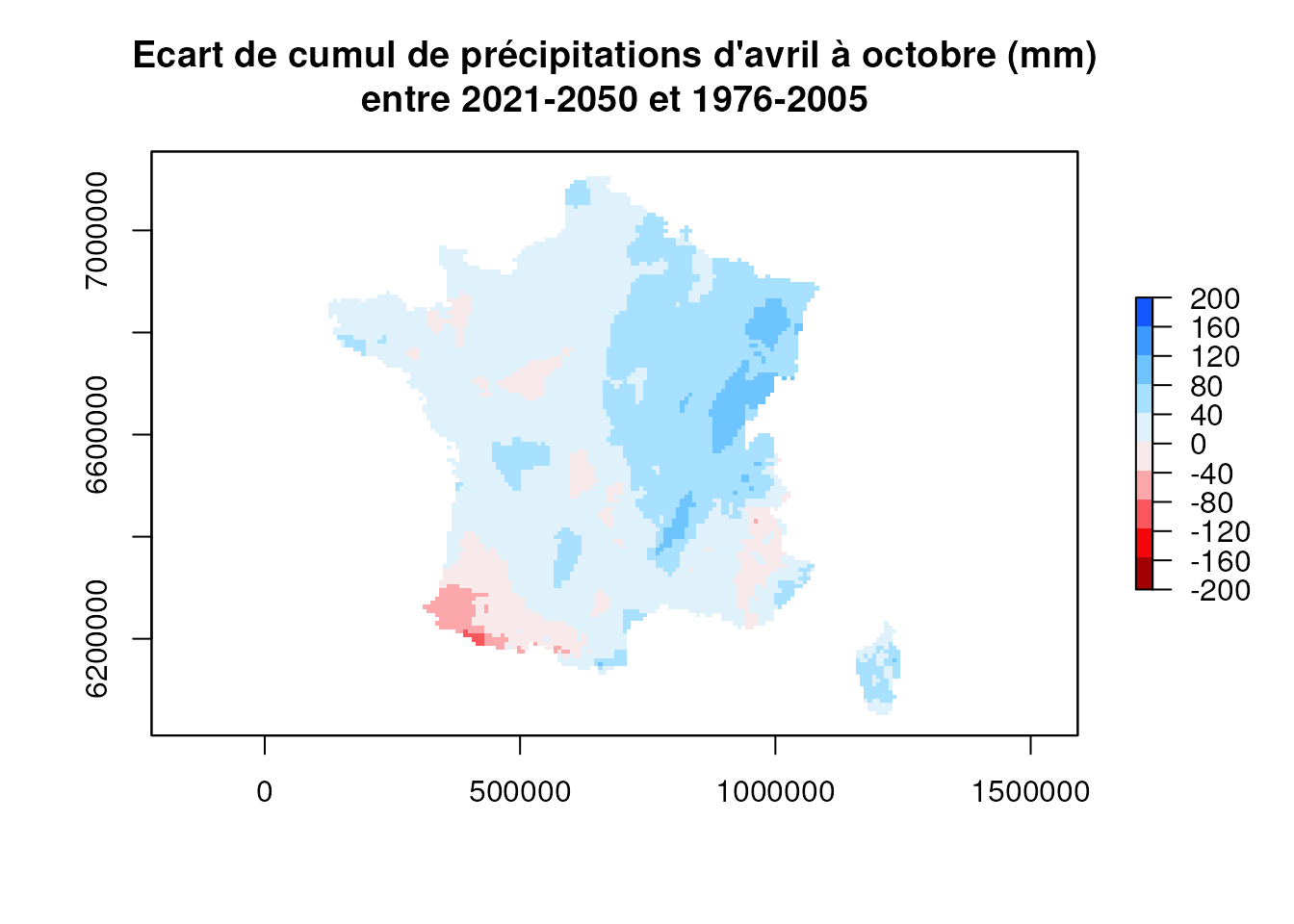
On souhaite tout d’abord visualiser les données DRIAS. On peut passer par les données sauvegardées au format raster qui sont stockées dans l’espace de stockage du SSP Cloud. Ces données correspondent à des prévisions à horizon proche (2021-2050) dans un scénario sans réduction des gaz à effet de serre, la période de référence étant 1976-2005. Le code ci-dessous permet de récupérer les données.
drias_raster <- s3read_using(
function(f) readAll(brick(f)),
object = "2023/sujet2/diffusion/resultats/drias.tif",
bucket = "projet-funathon",
opts = list("region" = ""))
drias_df <- as.data.frame(drias_raster, xy = TRUE) %>% tidyr::drop_na()
colnames(drias_df) <- c(
"x",
"y",
"NORRRA",
"NORSTM6",
"NORSTM0",
"NORSDA",
"NORDATEVEG",
"NORDATEDG",
"NORDATEPG",
"ARRA",
"ASTM6",
"ASTM0",
"ASDA",
"ADATEVEG",
"ADATEDG",
"ADATEPG",
"ALTI"
)
drias_df %>% head(10) %>% kable()| x | y | NORRRA | NORSTM6 | NORSTM0 | NORSDA | NORDATEVEG | NORDATEDG | NORDATEPG | ARRA | ASTM6 | ASTM0 | ASDA | ADATEVEG | ADATEDG | ADATEPG | ALTI |
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 633222.7 | 7102519 | 456.2780 | 1922.019 | 3166.208 | 1.970000 | 37.00000 | 246.0152 | 155.0000 | 39.49532 | 186.3507 | 242.3591 | 0.1700000 | -6.000000 | -13.03036 | 9.924110 | 2.000000 |
| 641222.7 | 7102519 | 443.9634 | 1991.784 | 3237.846 | 1.703987 | 37.00000 | 231.2215 | 156.9705 | 35.80390 | 182.8417 | 240.3598 | 0.1305907 | -6.000000 | -15.95570 | 1.132903 | 2.000000 |
| 649222.7 | 7102519 | 443.9969 | 1995.343 | 3242.508 | 1.867559 | 37.00000 | 227.0574 | 157.9856 | 31.55116 | 181.4101 | 241.3846 | 0.3665546 | -6.000000 | -21.91386 | 1.985644 | 1.014356 |
| 657222.7 | 7102519 | 436.6389 | 2002.257 | 3246.031 | 2.067211 | 37.00000 | 232.9163 | 158.0000 | 24.69242 | 188.7246 | 250.9371 | 0.4686055 | -6.000000 | -18.05578 | 6.930274 | 2.521252 |
| 665222.7 | 7102519 | 435.9542 | 2000.975 | 3246.324 | 2.058347 | 37.00000 | 230.7862 | 158.4427 | 24.02779 | 186.6543 | 248.8595 | 0.4583470 | -6.000000 | -17.55724 | 3.799755 | 2.349262 |
| 673222.7 | 7102519 | 435.9155 | 2000.556 | 3245.985 | 2.050125 | 37.00000 | 230.5039 | 158.4992 | 23.95599 | 186.3690 | 248.5641 | 0.4501251 | -6.000000 | -17.50078 | 3.503910 | 2.497654 |
| 609222.7 | 7094519 | 479.2000 | 1851.100 | 3046.770 | 1.700000 | 39.00000 | 242.0000 | 152.0000 | 52.62000 | 193.7900 | 246.7000 | 0.0300000 | -8.000000 | -21.00000 | 6.000000 | 66.000000 |
| 617222.7 | 7094519 | 468.7802 | 1908.548 | 3134.198 | 1.729771 | 38.00763 | 244.9771 | 152.9924 | 51.21084 | 195.0900 | 248.3969 | -0.0692365 | -7.007635 | -17.03054 | 6.000000 | 20.351189 |
| 625222.7 | 7094519 | 461.3004 | 1925.008 | 3168.732 | 1.968268 | 37.00722 | 246.9856 | 154.9856 | 45.71983 | 190.3150 | 242.9796 | 0.1682681 | -6.007217 | -15.01443 | 5.007217 | 1.642246 |
| 633222.7 | 7094519 | 456.2343 | 1922.001 | 3166.199 | 1.970000 | 37.00000 | 246.0068 | 155.0000 | 39.44270 | 186.3171 | 242.3540 | 0.1700000 | -6.000000 | -13.01360 | 9.966008 | 1.996718 |
Les variables disponibles dans les données DRIAS sont systématiquement calculées sur la période 2021-2050 (préfixe NOR), et en écart avec la période de référence (préfixe A) :
- RRA : cumul de précipitations d’avril à octobre (mm)
- STM6 : somme de température base 6°C d’avril à octobre (°C)
- STM0 : somme de température base 0°C d’octobre (année-1) à juillet (année) (°C)
- SDA : nombre de jours d’été d’avril à juin (jour(s))
- DATEVEG : date de reprise de végétation en jour julien (date)
- DATEDG : Date de dernière gelée avec 1er juillet comme référence (date)
- DATEPG : Date de première gelée avec 1er juillet comme référence (date)
On souhaite visualiser la variable ARRA, qui correspond à la huitième couche du raster et correspond à l’écart de cumul de précipitations d’avril à octobre en mm. Pour faire cela, on pourra récupérer spécifiquement la couche désirée lors de l’utilisation de la fonction raster::raster. Pour la visualisation, on pourra utiliser raster::plot.
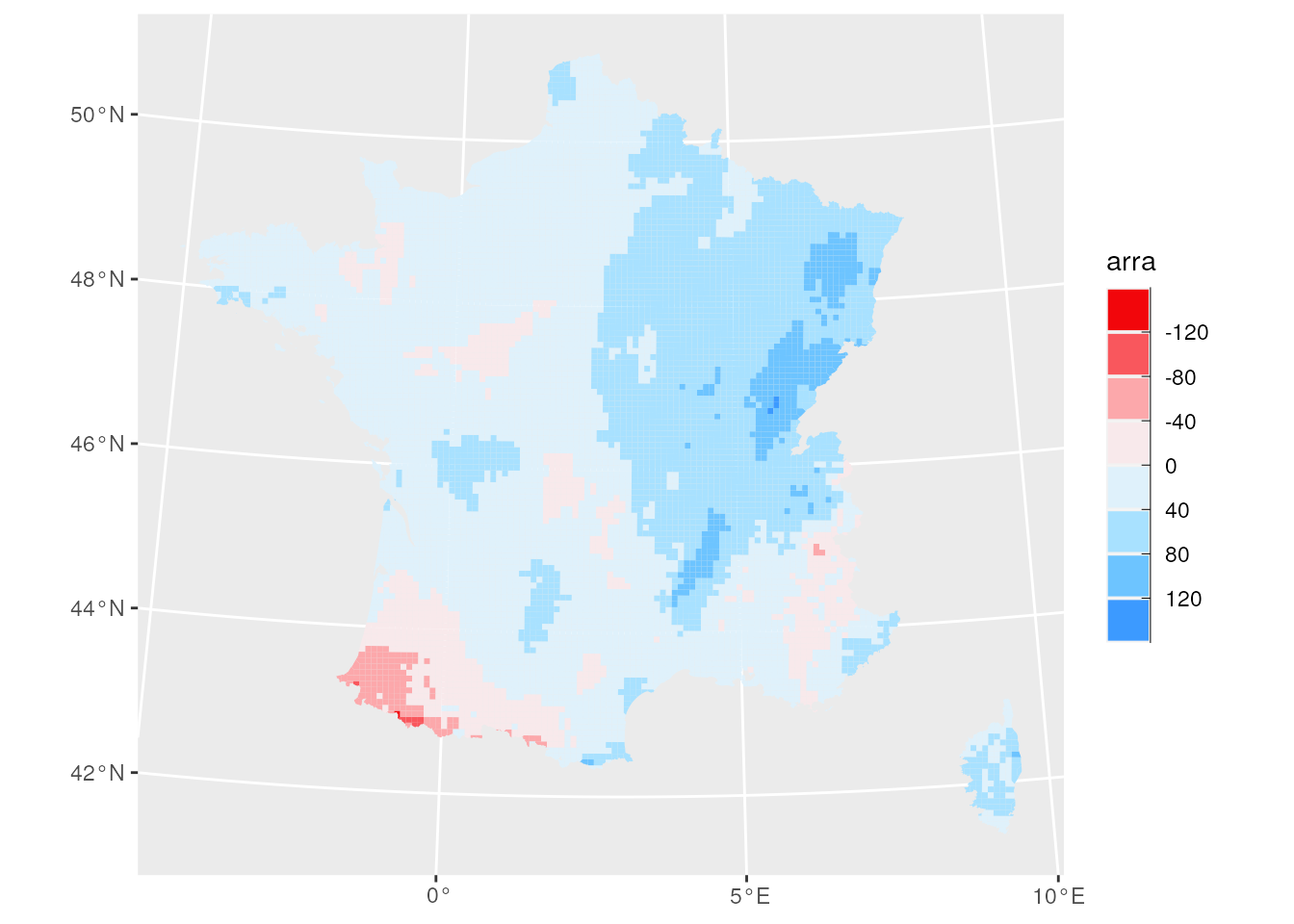
Sur la période 2021-2050, les précipitations vont augmenter presque partout, sauf dans le Sud-Ouest de la France.
3.3 Requêtes PostgreSQL
Les données DRIAS sont également stockées dans une base PostgreSQL qu’il est possible de requêter. La table drias.previsions contient une grille équivalente aux données raster précédemment utilisées. On souhaite obtenir la même visualisation que celle obtenue précédemment en requêtant la base de données. Pour ce faire, utiliser la fonction sf::st_read avec une requête sur la table drias.previsions. Pour la création de la carte, on peut utiliser le package ggplot2 et sa fonction geom_sf.
3.4 Appariement spatial entre données DRIAS et RPG
On aimerait associer chaque culture à une évolution en terme de cumul de précipitation. Pour ceci il faut tout d’abord récupérer pour chaque culture la surface des parcelles existantes dans chaque carreau de la grille DRIAS. Pour ceci on peut procéder à un appariement spatial des tables drias.previsions et rpg.parcelles de la base de données. On pourra s’aider de cette page de documentation, et utiliser ST_Intersects.
A ce stade, on a récupéré une table avec la surface par type de culture pour chaque carreau de la grille.
3.5 Calcul d’indicateurs par type de culture
On peut maintenant calculer un écart moyen de cumul de précipitation (d’avril à octobre) par unité de surface pour chaque culture, afin identifier celles qui seront impactées par des baisses de précipitations à horizon proche. Un fichier .csv contenant les intitulés complets des différentes cultures est disponible sur MinIO (bucket "projet-funathon", objet "2023/sujet2/diffusion/ign/rpg/CULTURE.csv").
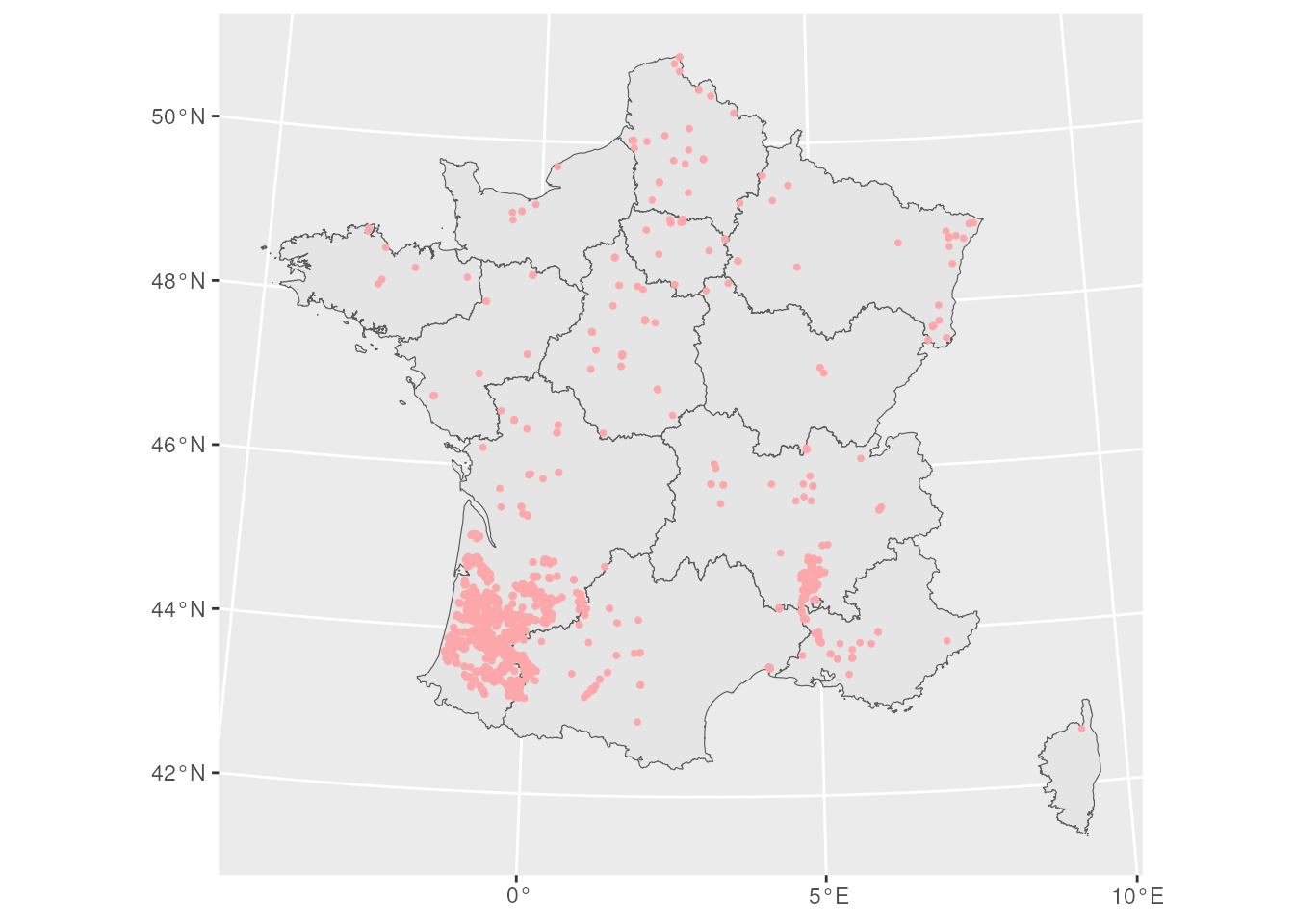
On observe que maïs doux est une culture qui va connaître une baisse de cumul de précipitations à horizon proche. Où sont situées les parcelles de maïs doux ? On souhaite dessiner une carte indiquant l’emplacement de ces parcelles. Pour ce faire, faire une requête de la table rpg.parcelles et utiliser ggplot2 et sa fonction geom_sf pour tracer la carte. Pour que les parcelles soient visibles, on pourra artificiellement augmenter leur surface grâce à la fonction st_buffer.
On observe comme attendu beaucoup de parcelles dans le Sud-Ouest où des baisses de précipitations sont attendues à horizon proche.
3.6 Pour aller plus loin
Pour aller plus loin, plusieurs idées :
- Calculer des écarts en pourcentage par rapport au niveau sur la période de référence pour avoir une idée plus parlante de la diminution ou de l’augmentation du cumul de précipitations;
- Regarder plutôt les prévisions climatiques à long terme, en utilisant la table
drias.previsions_hlqui concerne la période 2071-2100; - S’intéresser aux autres indicateurs qui existent dans les données DRIAS;